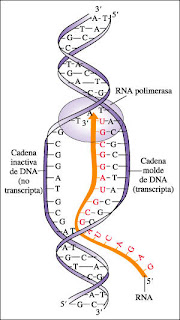
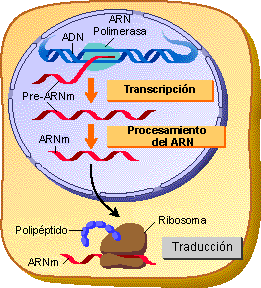
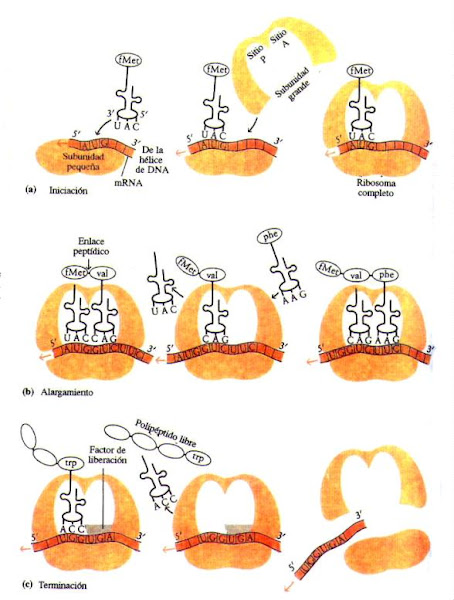
En 1955 Severo Ochoa descubrió y aisló una enzima de una célula bacteriana de Escherichia coli, que él denominó polinucleótido-fosforilasa y que luego fue conocida como ARN-polimerasa, cuya función catalítica es la síntesis de ARN (ácido ribonucleico), la molécula necesaria para la síntesis de proteínas.
En 1955 Severo Ochoa descubrió y aisló una enzima de una célula bacteriana de Escherichia coli, que él denominó polinucleótido-fosforilasa y que luego fue conocida como ARN-polimerasa, cuya función catalítica es la síntesis de ARN (ácido ribonucleico), la molécula necesaria para la síntesis de proteínas. Con esa enzima, Ochoa consiguió por vez primera la síntesis del ARN en el laboratorio, a partir de un sustrato adecuado de nucleótidos (sus componentes elementales). Un año más tarde, el bioquímico norteamericano Arthur Kornberg, discípulo de Ochoa, demostró que la síntesis de ADN también requiere otra enzima polimerasa, específica para esta cadena. Ambos compartieron el Premio Nobel de Fisiología y Medicina de 1959 por sus descubrimientos.
Estos extraordinarios hallazgos permitieron posteriormente el desciframiento del código genético (que se comprobó era universal para todos los seres vivos) y la confirmada capacidad reproductiva de los ácidos nucleicos hizo que éstos fueran ya considerados como las moléculas de la herencia biológica.Posteriormente, vista la importancia biológica de la doble hélice de ADN, Watson y Crick compartieron el Premio Nobel de Fisiología y Medicina de 1963. Severo Ochoa continuó investigando el mecanismo molecular de la lectura del mensaje genético y su expresión. En 1971 fue nombrado Director del Laboratorio de Biología Molecular de la Universidad Autónoma de Madrid. Dejó la Universidad de Nueva York en 1975, regresó a su país de origen y en la década de 1980 dirigió dos grupos de investigación en biosíntesis de proteínas simultáneamente, uno en el Instituto de Biología Molecular de Madrid y otro en el Roche Institute of Molecular Biology de Nueva Jersey, en Estados Unidos, hasta que en 1985 fijó su residencia definitivamente en España. Aunque se jubiló oficialmente en 1975, nunca abandonó la investigación.
En mayo de 1986 murió su mujer, y ello supuso para Severo un golpe muy duro que le sumergió en una especie de profunda depresión. A partir de entonces, Ochoa decidió no volver a publicar ningún trabajo científico más, con lo que puso totalmente fin a su brillante carrera. En 1993 murió en Madrid, a la edad de 88 años, a consecuencia de una neumonía.